ustas - stock.adobe.com
IA : comment générer de l’ADN avec des LLMs
Les grands modèles de langage (LLM) ont des applications potentielles qui vont au-delà des résumés ou des brouillons de mails. Ils peuvent aussi être utilisés pour créer des séquences synthétiques d’ADN.
Lorsque l’on parle de grands modèles de langage (LLM) de l’IA générative, les cas d’usages les plus souvent évoqués sont le résumé de texte, la génération de texte ou la réponse à des questions (comme avec ChatGPT). Mais ce n’est que la partie émergée de l’iceberg. Dans cet article, nous allons par exemple aborder la création de « phrases » cohérentes – de très longues phrases comportant des millions de lettres – dans une langue particulière qui s’appuie sur un alphabet inhabituel.
Cet alphabet n’est composé que de 4 lettres : A, C, G, T. Chacune représente une protéine : adénine (A), cytosine (C), guanine (G) et thymine (T). Vous l’avez deviné, c’est le langage de l’ADN, et les longues phrases sont des séquences d’ADN.
Schémas récurrents identifiés dans les séquences d’ADN
Exactement comme en français ou en anglais, les suites de lettres et de mots ne sont pas aléatoires (ce n’est pas du pur hasard). Certaines combinaisons de lettres se produisent fréquemment, d’autres jamais.
Les combinaisons rares peuvent indiquer une condition génétique particulière – comme les fautes de frappe dans la langue anglaise. Mais la grande différence est l’absence de séparateurs (virgules, espaces, points d’interrogation), ce qui crée de longues séquences ininterrompues de lettres. Dans ce contexte, un mot est une combinaison de deux, trois ou plusieurs lettres consécutives. Et tous les mots se chevauchent.
D’une certaine manière, la synthèse de l’ADN est, sur le papier, un peu plus facile que la génération d’un texte en français. Mais, il y a des autocorrélations à très grande échelle (long-range autocorrelations) et des règles non probabilistes.
Par exemple, comment et où insérer le « mot » qui identifie le sexe d’un être humain ? Nous ne répondrons pas à ces questions ici. Nous allons plutôt nous concentrer sur des schémas récurrents et des motifs à « petite échelle ». Ce qui, en fin de compte, n’est pas très différent de ce que font les LLM classiques, qui se concentrent principalement sur les tokens adjacents et les processus autorégressifs de prédiction.
Et dans les deux cas, l’embedding des mots est un élément essentiel.
La figure 1 montre trois sous-séquences d’ADN : une réelle, une synthétique et une aléatoire de lettres A, C, G, T. Pouvez-vous identifier des structures dans les deux sous-séquences du haut ?
 Figure 1 – Trois séquences d’ADN (réelle, synthétique, aléatoire)
Figure 1 – Trois séquences d’ADN (réelle, synthétique, aléatoire)
Données d’entraînement
Le data set d’entraînement se compose d’un certain nombre de séquences d’ADN humain. Il est accessible publiquement.
Le projet initial consistait à classer différentes sous-séquences présentant différents traits génétiques, sur la base des motifs récurrents (patterns) et des mots trouvés, y compris leurs distributions statistiques.
J’ai regroupé les données, et produit une séquence de quelques millions de lettres. En plus des quatre lettres A, C, G, T, on trouve un certain nombre d’occurrences de la lettre N, qui représente probablement des données manquantes.
Vous pouvez trouver ces données et le code Python sur mon dépôt GitHub. Vous pouvez y accéder en téléchargeant le document technique #34, ici.
Au passage, une question éthique intéressante concerne la protection de la vie privée : les sous-séquences individuelles pourraient permettre d’identifier des personnes en les comparant à des bases de données d’ADN existantes, et donc les maladies dont ces personnes pourraient souffrir. Heureusement, l’ADN synthétique rend impossible cette rétro-ingénierie. Ce qui est un de ses intérêts.
Notons que la technologie évoquée ici fonctionne également dans d’autres contextes. Par exemple, pour synthétiser des médicaments ou des molécules.
Synthétiser des séquences d’ADN
L’algorithme comporte deux étapes. Tout d’abord, il examine les paires de mots adjacents et calcule les occurrences et les probabilités conditionnelles. Ensuite, il génère séquentiellement de nouveaux mots sur la base du dernier mot généré et des probabilités en question. Finalement, vous obtenez un grand tableau d’embeddings, chaque embedding ayant des centaines ou des milliers de composants (mots apparentés), tout comme dans le LLM standard.
Plus précisément, le fonctionnement est le suivant.
Examinez un « mot » d’ADN S1 composé de n1 lettres consécutives, afin d’identifier les candidats potentiels pour le mot suivant S2 composé de n2 symboles. Attribuez ensuite une probabilité à chaque mot S2 en fonction de S1, et utilisez ces probabilités de transition pour échantillonner S2 en fonction de S1, puis déplacez-vous vers la droite de n2 lettres, recommencez, et ainsi de suite.
Vous finissez par construire une séquence synthétique de longueur arbitraire. Il existe une certaine analogie avec les chaînes de Markov et les processus autorégressifs.
Une application intéressante consiste à générer des séquences synthétiques – donc des êtres humains artificiels – puis à produire des images des personnes artificielles en question sur la base de leur ADN artificiel. Pour ce faire, on peut inférer des apparences physiques à partir de l’ADN : couleur des yeux et des cheveux, forme du nez, etc.
Évaluer la qualité de l’ADN synthétique
Pour évaluer les résultats, on peut comparer les fréquences d’un grand nombre de mots dans une séquence synthétique et dans une séquence réelle.
J’ai utilisé la distance de Hellinger, qui donne une valeur entre 0 (meilleur) et 1 (pire). La figure 2 est un diagramme Quantile-Quantile (QQ). Chaque point représente un mot. L’axe X représente la fréquence du mot en question dans les données synthétiques ; l’axe Y, sa fréquence dans l’ADN réel.
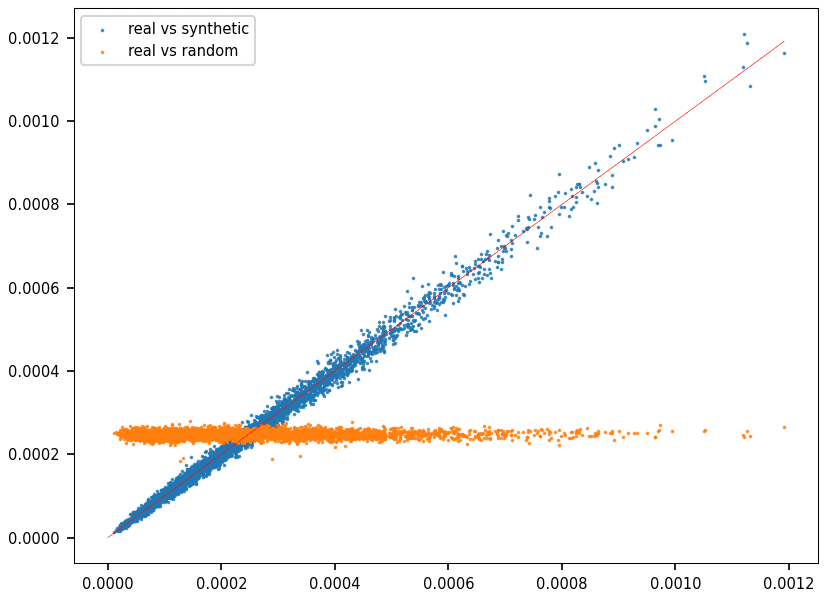 Figure 2 – Diagrame QQ : séquence aléatoire (orange), synthétique (bleu) et d’ADN réel (diagonale rouge)
Figure 2 – Diagrame QQ : séquence aléatoire (orange), synthétique (bleu) et d’ADN réel (diagonale rouge)
Il est clair, en regardant la figure 2, que les séquences d’ADN sont tout sauf aléatoires. L’ADN synthétique reproduit la distribution correcte, alors que l’ADN aléatoire est complètement à côté de la plaque.
Cependant, l’évaluation dépend de la longueur des mots utilisés. Dans ce cas, les 4 000 mots sélectionnés comportaient tous 6 lettres. Un graphique QQ avec dix mille mots de 8 lettres est présenté dans l’article n° 34 cité ci-dessus. La correspondance n’est pas aussi bonne que dans la figure 2.
En règle générale, ces diagrammes QQ ne permettent pas de rendre compte des interactions à très grande échelle.